L'échec de la théorie de l'évolution et l'arbre de la vie
READINGTIME 📃 Temps de lecture 11 minutes
Depuis les années 90, la crédibilité de la théorie de l'évolution en tant qu'hypothèse scientifique pour expliquer l'origine et l'évolution du vivant décroît significativement.
Cet article est consacré au concept de tree of life (arbre des espèces ou arbre de la vie). Nous allons passer en revue plusieurs études récentes, et montrer que la comparaison des génomes entre les espèces et les groupes d'espèces n'a pas permis de les réunir au sein d'un arbre de la vie. Les biologistes évolutionnistes s'attendaient à pouvoir fixer mathématiquement les relations de parenté entre les espèces. Les résultats des séquençages ADN pointent dans la direction opposée, à savoir des relations qui n'ont aucun sens vis-à-vis du paradigme darwinien de génération avec modification.
QUOTE«[Le projet d'arbre des espèces] est dans un état désastreux, taillé en pièces par une armée d'indications négatives» (3)
QUOTE«De nombreux biologistes soutiennent désormais que le concept d’arbre est obsolète et doit être abandonné» (3)
QUOTE«Nous avons détruit l'arbre des espèces. Il ne s'agit plus d'un arbre, la topologie est entièrement différente. Qu'aurait pensé Darwin de ces découvertes ?» (4)
Commençons par introduire brièvement la théorie de l’évolution.
D'après la théorie de l'évolution, toutes les espèces vivantes, et a fortiori tous les animaux sont reliés par la généalogie, ils partagent tous un ancêtre commun. Deux phénomènes sont invoqués pour expliquer l'apparition des espèces, le premier est celui des mutations génétiques, le second celui de la sélection naturelle.
Les étapes de l’évolution:
- (1) Une ou plusieurs mutations, donc des erreurs de copie du génome surviennent sur un individu, la conséquence est un changement au niveau phénotypique (ça peut être musculaire, osseux, ou alors au niveau des capacités cognitives etc…)
- (2) La/les mutation(s), et donc le phénotype nouveau est transmis à la descendance.
- (3) Ce phénotype nouveau doit ensuite être retenu par la sélection naturelle. D'une manière ou d'une autre les individus avec la mutation se séparent, se distinguent, dominent les autres ou ont en tout cas un rapport changé avec leur environnement. La conséquence de ce changement est une fixation de la ou les mutations dans le nouveau groupe. On parle alors d’une nouvelle espèce.
- (4) Éventuellement l'interfécondité disparaît progressivement ou brutalement entre deux populations avec un ancêtre commun.
Les points (2) et (3) peuvent être interchangés, peu de données sont précises dans la théorie de l'évolution, donc on peut imaginer que la sélection naturelle ait lieu avant la transmission des modifications et la formation d'une population mutante, ou alors après.
Les singes, les baleines, les koalas, les ornithorynques, les chameaux, les bactéries les papillons sont apparus selon ces mécanismes.
Nous avons arrondi et simplifié volontairement. Le paradigme est finalement facile à comprendre: une succession de micro-changements, qui finissent par distinguer radicalement une population d’une autre, aboutissant à une nouvelle espèce, ou une nouvelle famille.
Ce qui découle de ce paradigme, c’est la possibilité de représenter les espèces au sein d’un arbre, de les relier les unes aux autres. Toutes les espèces sont cousines avec les autres, elles sont reliées par des ancêtres communs, l'espèce ancêtre commun d'ailleurs peut avoir survécu ou n'avoir pas survécu. Dans le cas homme-chimpanzé, les évolutionnistes soutiennent que l'ancêtre commun n'a pas survécu, on suppose très fortement que c'est une espèce de type singe.
L’ADN est donc le support, le témoin de ces millions et millions d’années d’évolution, d’une espèce à l’autre, un même gène peut avoir perduré, en comparant ce même gène chez les deux espèces, nous pouvons éventuellement avoir un indice sur leur proximité. Nous avons un très bon élément pour valider ou invalider les prédictions de la théorie de l'évolution, pour valider ou invalider des parentés déterminées par comparaison anatomique.
C’est un peu comme quand on fait un test ADN pour savoir, les maths ne mentent pas. Nous pouvons établir avec certitude que tel enfant est ou n'est pas le fils de tel homme, même si le résultat est inattendu.
La construction des arbres phylogénétiques grâce à l'ADN
SMALLIMAGE 
Le but ici est de montrer comment les arbres phylogénétiques sont construits à partir de l’ADN. Ensuite nous passerons en revue plusieurs résultats.
Prenez un gène A chez une espèce 1, nous allons comparer ce gène A avec le même gène A chez d'autres espèces. Déjà il faut trouver des espèces qui ont un gène similaire, qui remplit la même fonction sinon la comparaison n'a pas de sens.
Une fois que vous avez identifié deux autres espèces, les espèces 2 et 3 par exemple, qui ont ce gène, vous pouvez comparer la séquence de nucléotides du gène A chez ces trois espèces.
Si vous obtenez 5% de différence entre l’espèce 1 et l'espèce 2, et 15% entre l’espèce 1 et l'espèce 3. Vous pouvez déduire que l'espèce 1 est plus proche de l'espèce 2 que de l'espèce 3. L’espèce 1 et l'espèce 2 ont un ancêtre commun plus récent que les espèces 1 et 3.
Vous pouvez dessiner un arbre de ce genre.

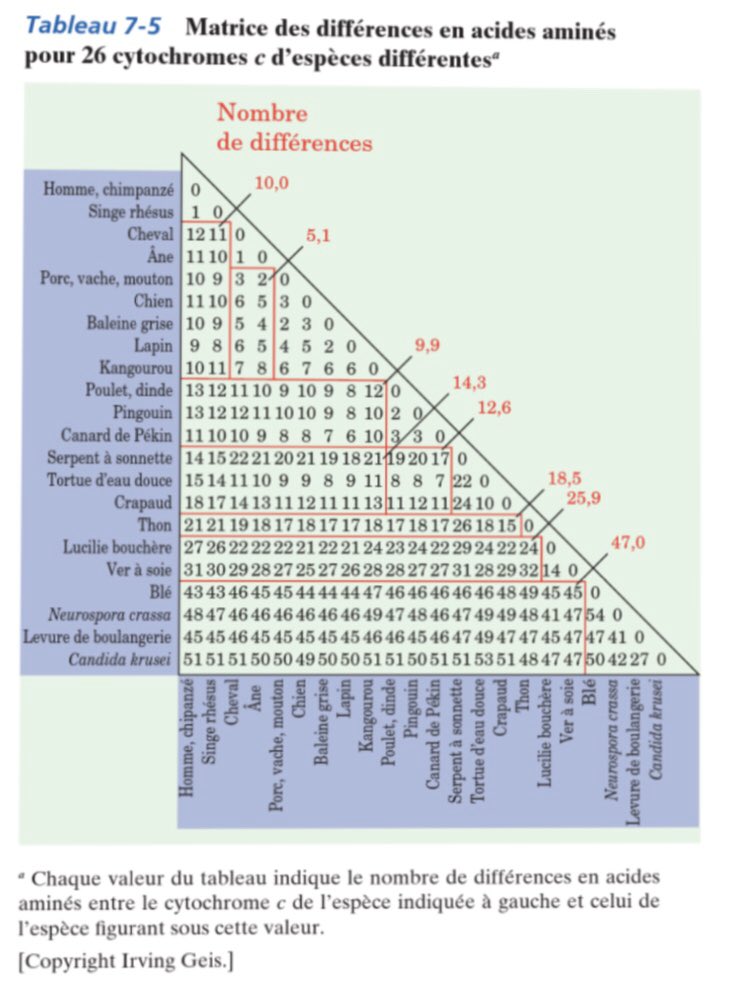
Dans le cas du gène qui code pour la protéine cytochrome C qui sert à la respiration cellulaire, nous obtenons des tableaux de ce genre, on appelle ça une matrice.
Maintenant reprenez vos espèces 1, 2 et 3, considérons un autre gène B. Nous obtenons 3% de différence entre les espèces 1 et 3, et 40% entre les espèces 1 et 2. Si on construit un arbre en se basant sur la proximité de ces trois espèces vis-à-vis du gène B, on obtient cet arbre.

Cela devient étrange, ou en tout cas inattendu, notre comparaison précédente indiquait que 1 et 2 ont un ancêtre commun proche, pour le gène B on devrait également trouver des séquences proches entre 1 et 2, par rapport à 1 et 3. Nous vennons de rencontrer une anomalie entre la phylogénie d’après le gène A et la phylogénie d’après le gène B.
Maintenant prenons un troisième gène C. Cette fois il n'y a que 2% de différence entre 2 et 3, et 30% de différence entre 1 et 2.
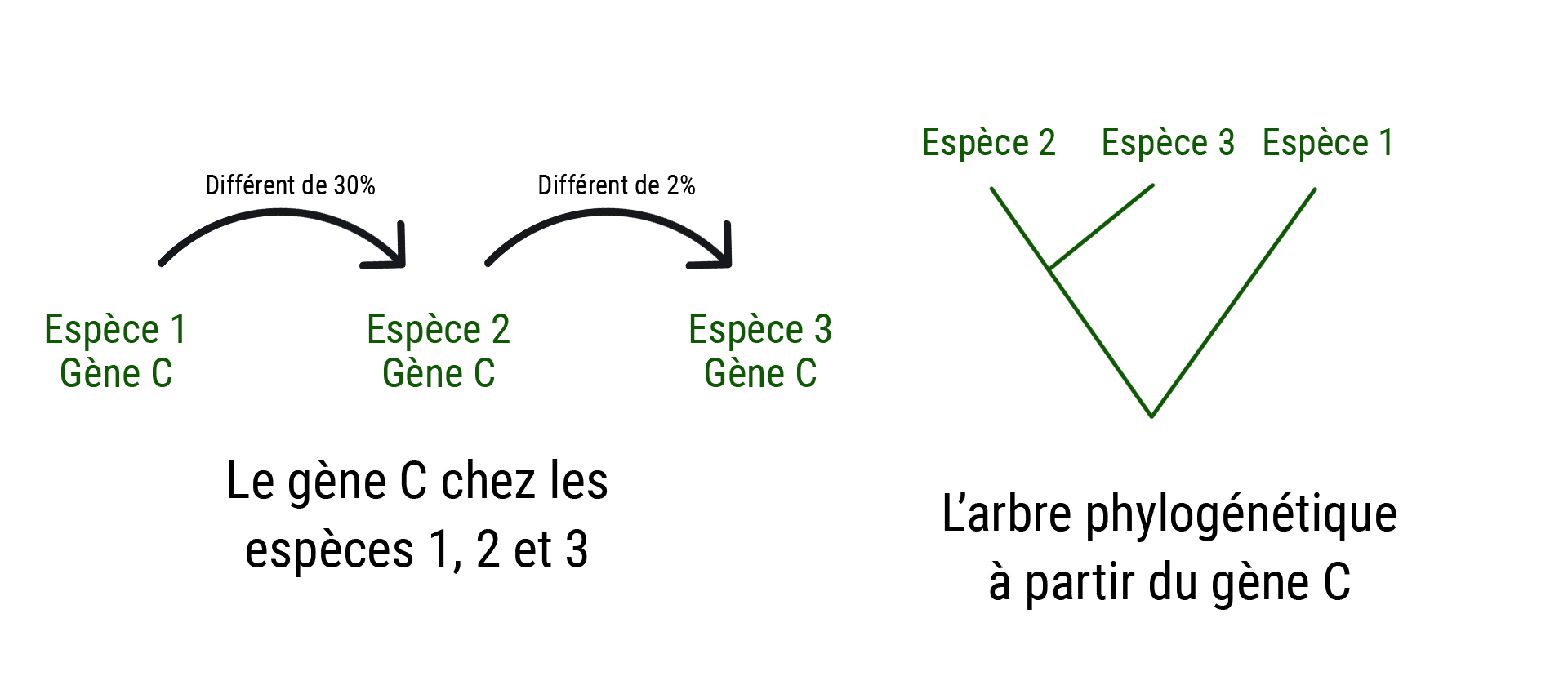
Notre arbre devient vraiment caduque, selon que l'on donne la priorité à tel ou tel gène, l'embranchement n'est pas au même endroit. Les données ne reflètent pas un héritage avec des modifications. Impossible de réunir les espèces dans un arbre.

Si les séquençages des gènes A B et C donnaient des proximités identiques, on aurait pu obtenir au contraire une phylogénie cohérente.
Des résultats difficiles à digérer
Dans la mesure où il est de plus en plus simple de séquencer des ADN, de plus en plus d'études sont faites pour déterminer les relations, ou non-relations entre les espèces.
A votre avis, les données ADN des différentes espèces et la comparaison des séquences de nucléotides sont-elles cohérentes, refletent-elles le paradigme darwinien de génération avec modification ?
Un point important, dans les études que nous listons ici, il peut s’agir de parentés entre deux espèces, comme hippopotame et baleine, parfois il s’agit des liens de parentés entre des groupes d’espèces, des familles, ou genres comme cétacés, pachydermes, ruminants, équidés etc.
QUOTESMALL«evolutionary trees from different genes often have conflicting branching patterns» (1)
QUOTE«Lorsqu’ils sont chacun basé sur un gène, les arbres reliant les espèces ont souvent des embranchements incohérents les uns avec les autres» (1)
QUOTESMALL«phylogenetic conflict is common, and frequently the norm rather than the exception» (2)
QUOTE«Les conflits en phylogénétique sont courants, ils sont la norme et non pas l'exception» (2)
QUOTESMALL«[The tree of life project] lies in tatters, torn to pieces by an onslaught of negative evidence» (3)
QUOTE«[Le projet d'arbre des espèces] est dans un état désastreux, taillé en pièces par une armée d'indications négatives» (3)
QUOTESMALL«Many biologists now argue that the tree concept is obsolete and needs to be discarded» (3)
QUOTE«De nombreux biologistes soutiennent désormais que le concept d’arbre est obsolète et doit être abandonné» (3)
QUOTESMALL«We have just annihilated the tree of life. It's not a tree anymore, it's a different topology [pattern of history] entirely. What would Darwin have made of that ?» (4)
QUOTE«Nous avon détruit l'arbre des espèces. Il ne s'agit plus d'un arbre, la topologie est entièrement différente. Qu'aurait pensé Darwin de ces découvertes ?» (4)
QUOTESMALL«A complete and accurate tree of life remains an elusive goal» (5)
QUOTE«Construire un arbre réunissant les espèces complet et cohérent est irréalisable» (5)
QUOTESMALL«certain critical parts of the TOL may be difficult to resolve, regardless of the quantity of conventionnal data available» (6)
QUOTE«Certaines parties critiques du tree of life (arbre de la vie) pourraient demeurer difficiles à résoudre, indépendament de la quantité de données disponible.» (6)
QUOTESMALL«many recent studies have reported support for many alternative conflicting phylogenies» (6)
QUOTE«De nombreuses études récentes supportent différentes phylogénies en conflit les unes avec les autres» (6)
QUOTESMALL«a 50-gene data matrix does not resolve relationships among most metazoan phyla» (7)
QUOTE«Notre matrice de 50 gènes ne résout pas les relations entre la plupart des phylas métozoaires"» (7)
Le terme "Phyla métozoaires" est équivalent à "genres animaux"
QUOTESMALL«despite the amount of data and breadth of taxa anlyzed, relationships among most metazoan phyla remained unresolved» (7)
QUOTE«Malgré la quantité de données, les relations entre les genres animaux demeurent non-résolues"» (7)
QUOTESMALL«So, complex genealogical problems often remain complex even after using genomic data. We haven’t "solved" any of the so-called genealogy problems, we have simply made clear in what way they are complex. That is, genomics data generally reveal reticulate evolutionary histories, not simple tree-like ones.» (9)
QUOTE«Les problèmes de généalogie demeurent complexes même avec les séquençages d'ADN. Nous n'avons pas "résolu" un seul des problèmes de généalogie, nous avons simplement mis en évidence leur complexité. Les séquençages et comparaisons ADN révèlent un schéma d'enchevetrement / de noeuds, au lieu d'un arbre.» (9)
QUOTESMALL«the genomics data make it clear that, while humans share slightly more genes with chimpanzees than with other great apes, we still share some with gorillas but not chimpanzees, and with orangutans but not either chimpanzees or gorillas. Also, chimpanzees share some genes with gorillas that we do not share. The situation is now clearer, but the tree incongruence remains.» (9)
QUOTE«Par rapport aux autres grand singes, les données ADN révèlent que c'est avec les chimpanzés que nous avons le plus de gènes en commun, cependant nous partageons des gènes avec les gorilles que n'ont pas les chimpanzés, nous partageons aussi des gènes avec les orang-outangs que les chimpanzés et gorilles n'ont pas. La situation est aujourd'hui plus claire, mais les incongruences de l'arbre demeurent.» (9)
Certain scientifiques proposent de revoir radicalement la représentation de type arbre, basée sur des bifurcations et isolations de branche, et poussent pour un modèle basé sur des réseaux, des noeuds et des transfers horizontaux fréquents.
QUOTESMALL«Non-tree-like phylogenetic results are currently not seen by most phylogeneticists as refuting the paradigm of a phylogenetic tree, but instead are the result of inadequate phylogenetic tree-models and/or insufficient data […] a shift to network thinking would embrace all of the tree results as well as the non-tree ones, and would thus form a viable new paradigm» (10)
QUOTE«La plupart des phylogénéticiens voient l'abolition du paradigme "arbre de la vie" comme une solution de secours aux phylogénies inadéquates et au manque de données ADN […] nous pensons qu'au contraire, transiter vers un modèle de réseaux [ou noeuds] permettrait d'avoir un nouveau paradigme qui engloberait les phylogénies de type "arbre de la vie" et les autres.» (10)
Comme cela est expliqué dans un autre article sur l’expérience de Lenski, chaque espèce a des gènes qui n’ont aucune similarité avec les gènes des autres espèces, chaque espèce est organisée uniquement. Il y a cependant des gènes qui sont partagés, que plusieurs espèces ont en commun.
Dance cette dernière étude une matrice de 50 gènes a été utilisée, les gènes ne sont pas choisis au hasard, évidemment il faut écarter les gènes orphelins, et prendre des gènes qui sont partagés ou semblables chez toutes les espèces étudiées.
Même avec cette présélection méticuleuse, ils ont été dans l'incapacité de classer les espèces dans un arbre. De dire qui descend de qui ou qui a un ancêtre commun avec qui.
Dans notre étude sur la convergence évolutive nous prenions l'exemple de la chauve-souris et du dauphin qui partagent un gène quasiment identique pour se situer dans l’espace grâce à des ondes haute-fréquence. Si on se basait uniquement sur ce gène pour dessiner notre arbre, la chauve-souris et le dauphin seraient très proches, ce qui évidement n'est pas logique, ces deux animaux sont trop différents. Ce genre de situation est ignoré dans cette dernière étude, cela donnerait des résultats encore plus incohérents. Les matrices sont donc faites à partir de gènes qui ont une répartition importante au sein des espèces.
Pour aller plus loin, nous vous invitons à lire les études qui sont listées en bas de la page. Des explications, ou des pistes sont parfois données pour expliquer les incohérences, et les conflits entre les différents arbres phylogénétiques, certain tentent d'expliquer cela en conservant une approche évolutionniste, d'autres ont simplement abandonné la notion d'arbre de la vie réunissant les famille animales.
Les concepts de convergence évolutive et transfert de gène horizontal sont très fortement liés à cette démarche, ça sera le thème d’une prochaine étude.
Le concept darwinien d'arbre des espèces, tree of life en anglais est petit à petit abandonné. Les séquençages ADN n'ont pas fait apparaître des relations de parenté logiques et cohérentes entre les espèces, entre les genres, entre les familles ou ordres. Au contraire, les séquençages et comparaisons génétiques ont fait apparaître l’invalidité des arbres et classifications évolutionnistes traditionnelles.
Dans certaines parutions scientifiques, vous verrez le terme tree (arbre en anglais) remplacé par le terme bush (buisson), par exemple dans cet article de 2006 de Antonis Rokas and Sean B Carroll. A défaut d'être en mesure de relier les grand groupes d'espèces les uns aux autres, plusieurs biologistes étudient les relations sur des groupes très restreints, sachant pertinemment qu'ils ne seront jamais en mesure de relier les bushes (buissons) entre eux.
Beaucoup de scientifiques ont aussi disqualifié le scénario darwinien et l’évolution, ils ne parlent donc ni de buissons ni d’arbre de la vie.
Il est impossible de dessiner un arbre regroupant les espèces animales, et d'avoir une cohérence entre les séquences ADN des espèces qui y figureront. Il n'y a pas quelques difficultés, ni quelques incohérences, c'est le modèle général darwinien ou néo-darwinien qui est faux. Il ne s’agit pas d’une ou deux études, mais de dizaines et dizaines d’études récentes, qui disent généralement la même chose.
Si l'on ajoute la surcouche des gènes orphelins, le paradigme darwinien est alors complètement atomisé.
Revenons à l’exemple de la protéine cytochrome C qui est souvent donné comme une preuve de l'évolution et de la cohérence des distances génétiques entre les espèces animales.
Oui, si l'on se concentre sur le gène qui code pour la protéine cytochrome C, on obtient des distances génétiques qui peuvent être une indication pour la validité de l’évolution. Il y a une correspondance générale avec l'arbre de la vie traditionnel, réalisé en comparant les anatomies des animaux. Mais si on prend un autre gène, on obtient un arbre en conflit avec le premier, et si on prend encore un autre gène on a encore une reconfiguration de la généalogie, et un conflit. Le gène cytochrome C est toujours mis en avant simplement car il est en relative adéquation avec les classifications des paléontologistes, les autres gènes qui donnent des résultats différents sont eux plutôt ignorés ou rangés dans la catégorie "on ne comprend pas".
Le point central est que si vous ne sélectionnez que les éléments qui prouvent une théorie, votre théorie sera prouvée et valide. Mais si vous voulez être vraiment sûr qu’elle est valide, soyez aussi en mesure d’analyser, de recevoir les éléments qui peuvent l’invalider.
La théorie de l’évolution est invalide, il y a tout simplement trop de contre-indications. Cet article ne s’est concentrée que sur les comparaisons des génomes.
Conclusion
Les espèces n'ont pas évolué et n’évoluent pas, il n'y a pas eu d'évolution inter-espèce. La science, le séquençage et la comparaison des génomes invalide radicalement la théorie de Darwin, et toutes les déclinaisons qui ont pu être faites au XXème et XXIème siècle. Les comparaisons d'ADN invalident le modèle darwinien basé sur la généalogie entre les espèces, l'évolution et la transmission des caractères.
A nouveau
Les espèces n'ont pas évolué et n’évoluent pas, il n'y a pas eu d'évolution inter-espèce. La science, le séquençage et la comparaison des génomes invalide radicalement la théorie de Darwin, et toutes les déclinaisons qui ont pu être faites au XXème et XXIème siècle. Les comparaisons d'ADN invalident le modèle darwinien basé sur la généalogie entre les espèces, l'évolution et la transmission des caractères.
Restez connecté sur niwrad.fr pour profiter d'articles sur le thème de la théorie de l'évolution confrontée aux avancées et découvertes scientifiques du XXIème siècle.
La rédaction
Références
(1) Degnan and Rosenberg, Department of Human Genetics, University of Michigan, "Gene tree discordance, phylogenetic inference and the multi-species coalescent", Trends in Ecology & Evolution Volume 24, Issue 6, June 2009, Pages 332-34
(2) Liliana M Dávalos, Andrea L Cirranello, Jonathan H Geisler, Nancy B Simmons, Department of Ecology and Evolution, State University of New York, "Understanding phylogenetic incongruence: lessons from phyllostomid bats"
(3) Lawton, G. 2009. Why Darwin Was Wrong About the Tree of Life. New Scientist. 2692: 34-39.
(4) Syvanen and Ducore "Whole genome comparisons reveals a possible chimeric origin for a major metozoan assemblage", Journal of Biological Systems Vol 18, June 2010
(5) Antonis Rokas, Université de Vanderbilt, molecular systemacist "Spotlight: drawing the tree of life" https://www.broadinstitute.org/news/spotlight-drawing-tree-life
(6) Antonis Rokas , Sean B Carroll, "Bushes in the tree of life", PLOS Biology November 14, 2006 https://journals.plos.org/plosbiology/article?id=10.1371/journal.pbio.0040352
(7) Antonis Rokas , Sean B Carroll, "Animal evolution and the molecular signature of radiations compressed in time", Science 23 Dec 2005: Vol. 310, Issue 5756, pp. 1933-1938 https://science.sciencemag.org/content/310/5756/1933
(8) Olivier Jeffroy, H. Brinkmann, F. Delsuc, H. Philippe, Phylogenomics: the beginning of incongruence? (2006) semanticscholar.org/paper/Phylogenomics%3A-the-beginning-of-incongruence-Jeffroy-Brinkmann
(9) David Morrison (Department of Organismal Biology , Sweden), Is phylogenomics tree-like? (2014) phylonetworks.blogspot.com/2014/10/is-phylogenomics-tree-like.html
(10) David Morrison (Department of Organismal Biology , Sweden), Conflicting placental roots: network or tree? (2013) phylonetworks.blogspot.com/2013/08/conflicting-placental-roots-network-or.html