L’impossibilité mathématique pour l'évolution humaine
READINGTIME 📃 Temps de lecture 8 minutes
Le but de cet article est d’expliciter les difficultés auxquelles est confrontée la chronologie évolutionniste traditionnelle, en un mot la séparation de deux lignées il y a 6 millions d’années, l’une aboutissant au chimpanzé moderne l’autre à l’homme moderne.
Nous nous appuierons principalement sur deux données, la première est la distance mathématique qui sépare l’ADN chimpanzé de l’ADN humain, la seconde est le taux de mutation par génération constaté par les médecins et chercheurs, connu et calculé précisément à partir des années 2010.
Chimpanzé et homme
Le chimpanzé est l'espèce animale la plus proche de l'homme sur le plan génétique.
Avant que le séquençage de l’ADN chimpanzé et humain soit fait, on l'avait déjà supposé, le chiffre qui faisait consensus autour de 2010 était de 35 millions de paires de base différentes entre humain est chimpanzé, entre 98% et 99% de similarités.
QUOTESMALL«About 35 million DNA base pairs differ between the shared portions of the two genomes, each of which, like most mammalian genomes, contains about 3 billion base pairs.»
QUOTE«Environ 35 millions de paires de base diffèrent entre les parties communes des deux génomes [humain et chimpanzé], ces génomes, comme ceux de la plupart des mammifères, sont longs de 3 milliards de paires de base environ.»
AUTHOR New Genome Comparison Finds Chimps, Humans Very Similar at the DNA Level, NIH News (National Institute of Health, Etats-Unis), 2005 (1)
Des chiffres plus récents donnent plutôt un pourcentage de similarités à 95% ou 96%.
QUOTESMALL«It is now clear that the genetic differences between humans and chimpanzees are far more extensive than previously thought; their genomes are not 98% or 99% identical”and “One consequence of the numerous duplications, insertions, and deletions, is that the total DNA sequence similarity between humans and chimpanzees is not 98% to 99%, but instead closer to 95% to 96%»
QUOTE«Il apparaît maintenant clairement que les différences génétiques entre humain et chimpanzé sont plus importantes que ce qui était admis, leur génome ne sont pas identiques à 98% ou 99%. […] La conséquence des duplications, insertions, deletions est que les similarités entre l’ADN humain et chimpanzé sont plutôt de 95% ou 96%.»
AUTHOR Todd M. Preuss, Human brain evolution: From gene discovery to phenotype discovery, PNAS, 2012 (2)
Preuss fait des assomptions évolutionnistes sur l’origine de ces différences, nous allons justement voir que ces assomptions ne s’accordent pas du tout avec les taux de mutations observés, qui sont beaucoup trop faibles. Basons nous donc sur ce chiffre de 96%, cela fait 128 millions de paires de base qui diffèrent sur 3,2 milliards. Dans la vidéo nous nous basons sur un chiffre beaucoup plus bas de 35 millions, qui correspond à l’ancienne estimation de 99% d’ADN en commun. La difficulté ressort pareillement avec un chiffre ou avec l'autre.
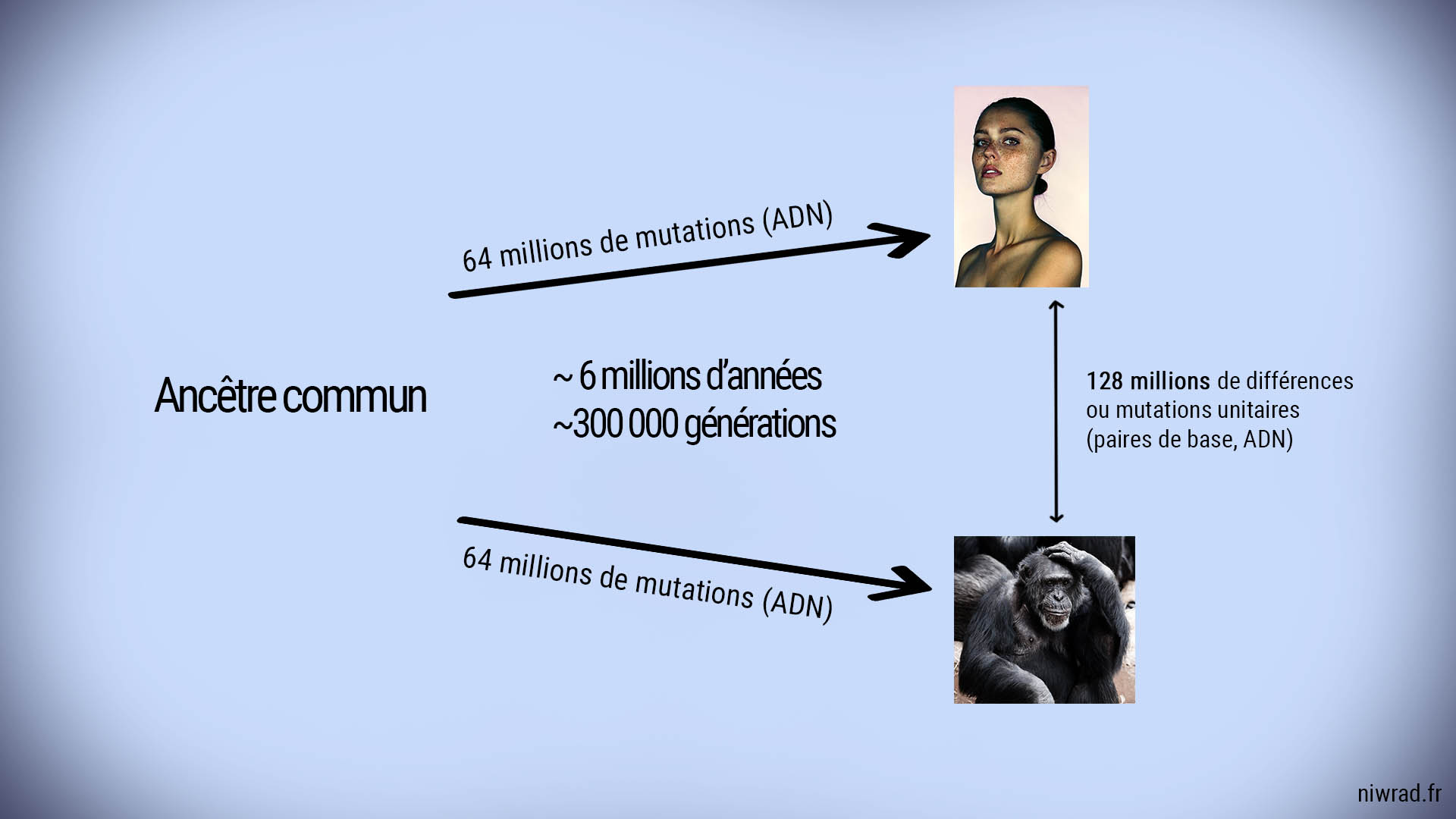
Plaçons nous dans le cadre de la théorie de l'évolution. Le chimpanzé est le plus proche cousin de l'homme, nous avons un ancêtre commun récent avec lui. Les évolutionnistes soutiennent que cet ancêtre commun a vécu il y a 6 millions d'années environ.
Il y a deux espèces, L'ADN diffère de 128 millions de caractères. On peut supposer que chaque branche a eu son lot de mutations. Cela fait 64 millions de modifications sur chaque branche, en 6 millions d'années, ayant eu pour résultat deux espèces distinctes.
Le taux de nouvelles mutations à chaque génération
On sait que les mutations s'accumulent dans l'ADN, pour toutes les espèces vivantes. Une étude récente qui fait plutôt consensus (Kong 2016) donne le chiffre de 1.29 × 10-8 mutations par position et par génération pour l'homme. Une deuxième plus récente donne quasiment le même chiffre.
QUOTESMALL«the average de novo mutation rate is 1.20×10−8 per nucleotide per generation.»
QUOTE«Le taux moyen de mutations de novo par nucléotide par génération est 1.20 × 10-8 [= 38 mutations pour un génome de 3,2 milliards pb].»
AUTHOR Augustine Kong, Michael L. Frigge, Gisli Masson, Soren Besenbacher, Patrick Sulem, Gisli Magnusson, Sigurjon A. Gudjonsson, Asgeir Sigurdsson, Aslaug Jonasdottir, Adalbjorg Jonasdottir, Wendy Wong, Gunnar Sigurdsson, G. Bragi Walters, Stacy Steinberg, Hannes Helgason, Gudmar Thorleifsson, Daniel F. Gudbjartsson, Agnar Helgason, Olafur Th. Magnusson, Unnur Thorsteinsdottir, and Kari Stefansson, Rate of de novo mutations and the importance of father’s age to disease risk, Nature, Août 2012 (3)
QUOTESMALL«we use 283 parent-offspring trios … We found 17812 SNVs, corresponding to a mutation rate of 1.29 × 10-8 per position per generation»
QUOTE«Notre étude se base sur 283 trios parents-enfant … Nous avons identifié 17812 SNV (PSN), correspondant à un taux de mutation de 1.29 × 10-8 [= 41 mutations pour un génome de 3,2 milliards pb] par position par génération.»
AUTHOR Søren Besenbacher, Patrick Sulem, Agnar Helgaso, Hannes Helgason, Helgi Kristjansson, Aslaug Jonasdottir, Adalbjorg Jonasdottir, Olafur Th Magnusson, Unnur Thorsteinsdottir, Gisli Masson, Augustine Kong, Daniel F Gudbjartsson, Kari Stefansson, Multi-nucleotide de novo Mutations in Humans, PLOS genetics, Novembre 2016 (4)
Gardons le chiffre de 41 mutations par génération en moyenne, il faut savoir que ce chiffre augmente avec l’âge des parents au moment de la conception.
Une base nucléique dans votre ADN correspond à une des 4 molécules A T G ou C, il y en a environ 3,2 milliards dans l'ADN humain. Votre enfant recevra en moyenne 41 erreurs de copie par rapport au mélange de votre ADN et de celui de votre compagne ou compagnon. Par exemple sur le chromosome 12, à telle localisation il y aura un G au lieu d'un C, éventuellement, la protéine codée par le gène pourra fonctionner moins bien, il pourra aussi n'y avoir aucune différence.
Une comparaison simple, est celle d’une feuille de papier, qui à chaque fois qu’elle est recopiée, est recopiée de manière inexacte avec 41 erreurs. Nous parlons pour l'ADN d’un alphabet à 4 caractères au lieu de 26.
A la génération n+2, il y aura en moyenne 82 nouvelles mutations qui se seront accumulées dans un génome, à la génération n+3, 123 nouvelles mutations, à la génération n+12, 492 nouvelles mutations etc.

Maintenant c'est très simple, partons de ce fameux ancêtre commun, qui a vécu il y a environ 6 millions d'années, si l’on prend un engendrement à 20 ans cela fait 300 000 générations.
300 000 (générations) x 41 (mutations par génération) = 12 300 000 mutations
Ce résultat est déjà inférieur aux 64 millions de modifications nécessaires pour expliquer la distance génétique entre nous et le chimpanzé. Il faut le multiplier par dix pour combler l’écart, si nous avions pris seulement 35 millions de différences homme-chimpanzé, il manquerait également 5 millions de mutations. Ce n’est malheureusement pas du tout le seul problème.
Quel est le second problème ? C'est celui du caractère aléatoire des mutations, en effet ces modifications ne suivent pas un chemin tout tracé qui va d'une espèce A à une espèce B, elles sont complêtement aléatoires.
C'est là qu’intervient la notion de sélection naturelle, la sélection naturelle est censée faire le tri parmi les mutants qui apparaissent, qu'il s'agisse d'un individu ou d'une population. C'est la sélection naturelle qui élimine les races ou espèces non-adaptées, et favorise justement celles qui s'adaptent et reçoivent la bonne somme de mutations dans le génome.
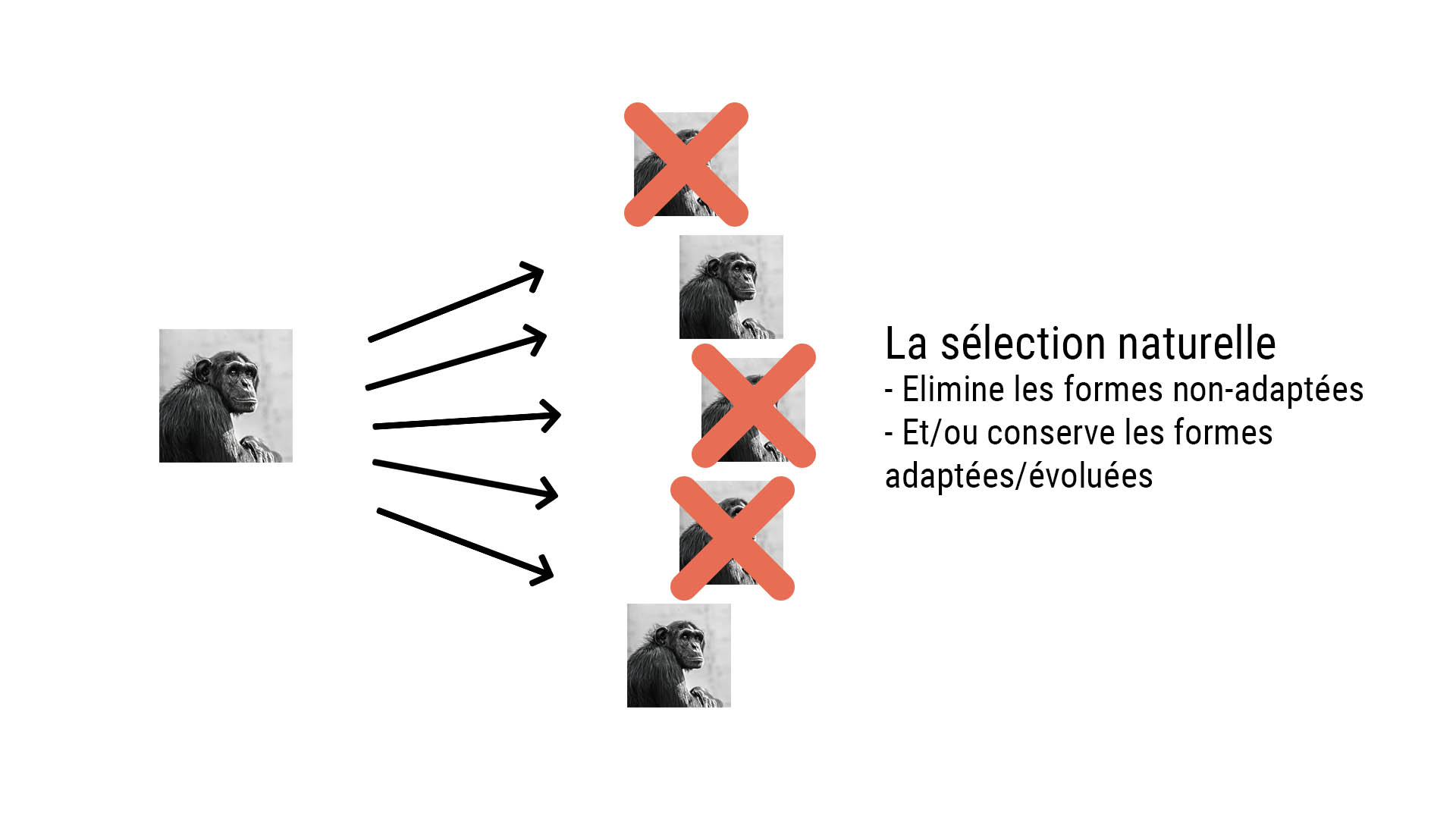
Quel est le problème titanesque que nous rencontrons maintenant ? Les mutations bénéfiques, qui apportent quelque chose de fonctionnel, qui construisent l'ADN au lieu de le détruire sont en fait très très très rares, nous ne sommes même pas sûr qu'elles existent vraiment.
Prenons la fameuse expérience de Lenski, après 30 000 générations, une mutation a désactivé un gène, et a permis à la bactérie E. Coli de consommer du citrate, obtenant ainsi, dans les circonstances de l'expérience, un avantage par rapport aux autres branches de E. Coli.
Gardons le chiffre d'une mutation bénéfique sur 1000, c'est déjà un chiffre très élevé et sûrement invalide. Considérons même que les autres 999 mutations sur 1000 sont neutres et ne sont pas nocives, nous savons que c'est faux également, il suffit de regarder la liste des maladies génétiques pour s'en convaincre.
Simulation
Nous sommes à la génération 0, je suis le singe (ou une population donnée et homogène de singes) ancêtre commun de l’homme et du chimpanzé. Statistiquement, combien faut-il faire d'enfants à la prochaine génération pour avoir au moins un enfant avec 20 ou plus de 20 mutations positives sur les 41 qu'il recevra ?
Nous ne préciserons pas les calculs ici, il faut probablement utiliser la fameuse loi de probabilité binomiale. Le chiffre est évidemment très très très élevé. Il faudrait 100 000, 1 million ou 10 millions d'enfants pour être sûr d'en avoir un qui va engendrer une population avantagée, une population évoluée.
Pour une population de 10 millions d’individus, si chaque couple fait 100 enfants, cela fait 500 millions d’individus à la génération suivante. Parmi ces individus il y en aura peut-être 4 ou 5 qui seront réellement évolués par rapport aux autres et qui constitueront des départs ou l’extension d’une branche d’évolution. Nous nous retrouvons quand même avec une masse asiatique d’individus avec une viabilité identique, ou alors légèrement avantagés ou même malades pour une minorité.
Pour expliquer par la théorie de l'évolution l'apparition de l'espèce humaine sur une branche, et des chimpanzés sur l'autre, avec un écart abyssal de 128 millions de nucléotides, et un taux de 41 mutations par génération, il faut deux éléments:
- Une quantité astronomique d'individus à chaque génération
- Une sélection naturelle très très forte qui conserve uniquement les individus adaptés, évolués. A savoir un individu sur 1000, 1 sur 2000, 1 sur 3000.
On voit bien que le premier point est déjà un problème, il n’y a aucun élément qui indique une population très importante dans un passé lointain, une majorité d’évolutionnistes considère même que la population humaine est restée stable, autour de quelques millions d’individus maximum sur des millions d’années. Quelques millions, cela ne suffit pas pour avoir une poignée d'individus qui se différencient grâce aux mutations bénéfiques, et survivent grâce à une sélection naturelle positive. Pour que la compétition et la sélection s’appliquent, il faut en plus d’une population importante, une proximité géographique, sans cela telle ou telle branche aurait simplement pu se déplacer et survivre sans forcément lutter pour des resources contre les autres races.
Rappelons une nouvelle fois qu'en environ 300 000 générations (ancêtre commun - homme moderne), il faut que plus de 60 millions de mutations se fixent dans le génome de la lignée humaine.
Pour le second point, le problème est donc l’absence totale de support expérimental (chez les mammifères en tout cas). Dans le cas où l'on obtiendrait quelques individus évolués à une génération donnée, il faut qu'ils prennent le dessus sur les autres, et se séparent d'une manière ou d'une autre, sans cela les mutations bénéfiques ne se fixeront pas dans une nouvelle branche (= population, race ou espèce), elles seront perdues.
Pour les singes par exemple, la sélection naturelle parfois peut faire un tri, certains individus vont être mis à l'écart d'un groupe, ou simplement ne pas survivre à cause d'une maladie mais c'est à peu près tout, il ne s’agit ici même pas d’une sélection positive, juste d’une élimination des moins aptes. A quel moment a-t-on observé qu’un avantage obtenu par une mutation ait permis à un groupe de mammifères de survivre et pas à un autre ? Même si un individu reçoit 41 bonnes mutations, court plus vite, est plus intelligent que les autres, ou bénéficie d'une transformation avantageuse pour telle ou telle articulation, cela ne va pas éliminer les autres pour autant.
Il y a des exemples de sélection naturelle négative chez les mammifères, à savoir une élimination ou non-reproduction des moins aptes, éventuellement suite à une ou plusieurs mutations (par exemple les maladies génétiques), cela ne permet aucunement une évolution. Par contre il n'y a pas d'exemple de sélection naturelle positive, à savoir la conservation d'un trait avantageux (musculaire, osseux, articulaire) obtenu suite à une ou plusieurs mutations aléatoires, et la séparation radicale d'un groupe par la sélection naturelle. C'est pourtant exactement l'évènement que la théorie prévoit, des dizaines de milliers de fois depuis la séparation des lignées homme et chimpanzé.
Troisième problème, si il y a effectivement eu toutes ces tentatives ratées, toutes ces branches, et seulement une branche ou deux qui s'en sont sorti, on devrait trouver des traces de ces hommes-singes, donc des squelettes par millions et millions. Nous vous invitons à consulter notre dossier sur l’évolution humaine pour constater que ces traces sont très fragmentaires.
QUOTE«Magré les fossiles trouvés, et les techniques d'analyse de ces cinquante dernières années, il n'y a toujours aucune hypothèse plausible pour l'origine du genre Homo [espèce humaine].»
AUTHOR Bernard Wood (George Washington University), Human Evolution; Fifty years after Homo habilis, Nature 508, 2014 (5)
Le registre fossile confirme exactement les données des biologistes, et les inférences que l'on peut faire à partir de ces données. Il ne révèle rien du tout, pas de formes transitionnelles, c’est logique, c’est simplement parce que ces formes transitionnelles n’ont pas existé.
Conclusion
Il n'y a pas d'ancêtre commun homme-chimpanzé, pas plus qu'il n'y aurait deux lignées d'évolution qui se seraient séparées il y a 6 millions d'années. Le caractère très improbable qu'a une somme de mutations bénéfiques d'apparaître à une génération donnée, et la probabilité encore plus faible que ce trait créé un avantage radical, et soit isolé par sélection naturelle rendent le scénario évolutionniste très improbable. Les contraintes des 300 000 générations (uniquement), et du très faible taux de mutations de novo par génération diminuent encore la plausibilité du modèle. Le modèle évolutionniste pour l'évolution humaine, la séparation et l'évolution des lignées homme et chimpanzé est bel et bien un récit, pas un modèle scientifique sérieux.
Avant les séquençages des ADN, on pouvait supposer que homme et singes étaient proches, qu'il n'y avait que 100, 1000, 10 000 modifications à apporter, sur 300 000 générations cela pouvait paraître plausible.
Il y a en fait une distance océanique de dizaines de millions de nucléotides entre le génome chimpanzé et le génome humain. Il n'y a que 41 mutations par génération en moyenne, et sur ces 41 mutations, une proportion énorme de mutations neutres ou nocives.
Le registre fossile, par la pauvreté des formes intermédiaires qu'il révèle confirme exactement l'impossibilité de l'évolution, qu'on peut deviner avec des calculs et des chiffres élémentaires sur le nombre de mutations par génération.
Nos calculs sont approximatifs, de nombreuses variables méritent d’être ajoutées, le raisonnement par contre est classique, on peut le trouver dans plusieurs autres articles scientifiques abordant la question de la distance génétique entre les espèces, en rapport avec la chronologie évolutionniste.
Nous avons centré notre article sur l’homme et le chimpanzé, il est probable que les mêmes impossibilités ressortiront pour d’autres familles animales. Nous aborderons dans un autre article le problème des gènes orphelins, qui est encore une surcouche aux difficultés exposées ici.
Restez connecté sur niwrad.fr pour profiter d'articles sur le thème de la théorie de l'évolution confrontée aux avancées et découvertes scientifiques du XXIème siècle.
La rédaction
Références
(1) New Genome Comparison Finds Chimps, Humans Very Similar at the DNA Level, NIH News (National Institute of Health, Etats-Unis), 2005 www.genome.gov/15515096/2005-release-new-genome…
(2) Todd M. Preuss, Human brain evolution: From gene discovery to phenotype discovery, PNAS, 2012 www.pnas.org/content/109/Supplement_1/10709.full
(3) Augustine Kong, Michael L. Frigge, Gisli Masson, Soren Besenbacher, Patrick Sulem, Gisli Magnusson, Sigurjon A. Gudjonsson, Asgeir Sigurdsson, Aslaug Jonasdottir, Adalbjorg Jonasdottir, Wendy Wong, Gunnar Sigurdsson, G. Bragi Walters, Stacy Steinberg, Hannes Helgason, Gudmar Thorleifsson, Daniel F. Gudbjartsson, Agnar Helgason, Olafur Th. Magnusson, Unnur Thorsteinsdottir, and Kari Stefansson, Rate of de novo mutations and the importance of father’s age to disease risk, Nature, Août 2012 www.nature.com/articles/nature11396
(4) Søren Besenbacher, Patrick Sulem, Agnar Helgaso, Hannes Helgason, Helgi Kristjansson, Aslaug Jonasdottir, Adalbjorg Jonasdottir, Olafur Th Magnusson, Unnur Thorsteinsdottir, Gisli Masson, Augustine Kong, Daniel F Gudbjartsson, Kari Stefansson, Multi-nucleotide de novo Mutations in Humans, PLOS genetics, Novembre 2016 journals.plos.org/plosgenetics/article?id=10.1371/journal.pgen.1006315
(5) Bernard Wood (George Washington University), Human Evolution; Fifty years after Homo habilis, Nature 508, 2014 nature.com/news/human-evolution-fi…